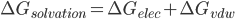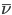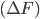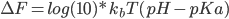*****2010 68,144miles FWD BLACK_PEARL NAVI 21800 21701 Ventura Blvd, Woodland Hills, CA 91364
****2010 51310miles AWD BLACK_parchment NAVI 24495 1332 PINE ST, Walnut Creek, CA 94596
***2013 51304miles FWD BROWN_beige dakota san jose 26500 (seen)
**2010 106909miles AWD Silver_tan NAVI san jose 18999
***2013 38120miles FWD GRAY_ALMOND BEIGE NAVI san jose 28900(seen)
***2010 87993miles FWD BLACK_LIGHT 19987 1500 West El Monte Way, Dinuba, CA 93618 (maybe on the way back)
****2012 105704miles FWD GOLD_BEIGE 19450 440 Fair Drive Suite 219, Costa Mesa, CA 92626 (gold color inside clean)
****2011 79731miles FWD gray_gray 24995 18820 Van Buren Blvd, Riverside, CA 92508 (inside clean)
****2012 47576miles FWD PEARL_LIGHT 26863 12747 Folsom Blvd, Folsom, CA (clean near)
****2010 51565miles FWD WHITE_GRAY NAVI 24945 69200 California 111, Cathedral City
****2011 74062miles AWD BLACK_LIGHT 23988 5860 Paradise Dr, Corte Madera, CA Marin Acura
***2010 76618miles AWD BLUE_LIGHT 23795 MODESTO (seen,screen yellow)
*****2010 44453miles FWD SILVER_BLACK 22997 2525 Wardlow Road Corona, CA, 92882 hyundai (low miles)
****2010 86372miles AWD GOLD_LIGHT 22988 ROSEVILLE CA (gold)
*****2011 50850miles FWD BLACK_BLACK 22900 2220 N Hollywood Way, Burbank, CA (low miles by appointment)
****2010 66765miles FWD WHITE_LIGHT 22400 464 W Main St,El Cajon,CA (had 2 accidents, color ok)
****2010 60291miles FWD Cerulean Blue Metallic_PARCHMENT NVAI 21988 807 E. Francisco Blvd, San Rafael, CA (BEEN THERE)
****2010 78684 miles AWD DARK BROWN_PARCHMENT NAVI 21988 12284 San Pablo Ave, Richmond, CA
****2011 63517miles FWD BLACK_IVORY 19995 700 South Brand Blvd, Glendale, CA (low price, not sure about NAVI)